Ha a Google mesterséges intelligenciája, a DeepMind ember lenne, az elmúlt években kétségtelenül jól szórakozott volna, ugyanis vég nélküli videójátékozással töltötte az idejét, és a gépi tanulás segítségével emberfeletti képességekre tett szert többek közt a StarCraftban és az Atari teljes játékkínálatában, valamint góban is a világ legjobb játékosává vált. A DeepMind alapítója és arca, Demis Hassabis persze mindig kiemelte, hogy az MI fő célja, hogy segítsen a világ megértésében, most pedig úgy tűnik, ennek is eljött az ideje, az AlphaFold nevű mesterséges intelligencia ugyanis képessé vált arra, hogy rövid idő alatt egy atomnyi hibahatárral megjósolja a fehérjék szerkezetét – írja az MIT Technology Review.
A DeepMind és a kilencvenes évek közepe óta futó Critical Assessment of protein Structure Prediction (CASP) nevű verseny szervezői hétfőn jelentették be, hogy az AlphaFold lényegében megoldott egy olyan problémát, ami már évtizedek óta foglalkoztatja a kutatókat, és az első mesterséges intelligencia lett, ami képes ilyesmire. Ahhoz, hogy megértsük a dolog jelentőségét, első körben érdemes kicsit beszélni arról, hogy miért olyan nehéz megállapítani a fehérjék struktúráját, illetve hogy pontosan mi a franc az a CASP.
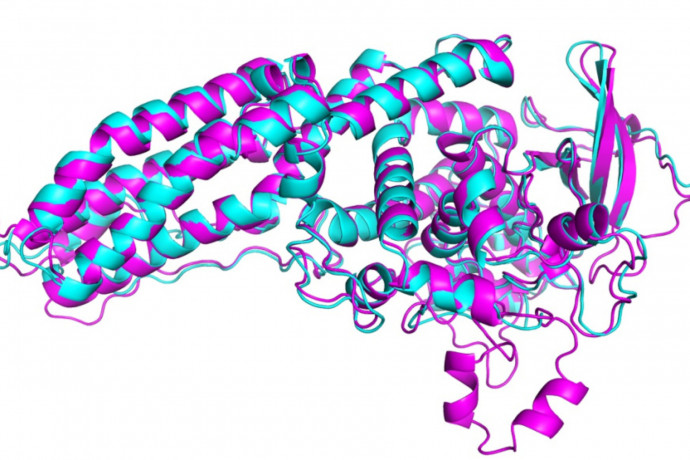
Egy fehérje tulajdonképpen nem más, mint egy aminosavakból álló, össze-vissza tekergő szalag, melynek funkcióját az határozza meg, hogy milyen a struktúrája, vagyis hogy pontosan hogyan tekereg. Ha megbízhatóan képesek vagyunk megállapítani a struktúrájukat, megérthetjük a működésüket, ezzel pedig közelebb kerülhetünk az élet alapvető mechanizmusainak feltárásához is. Ez ugyanakkor rendkívül nehéz feladat, hiszen a lehetséges struktúrák számában a határ a csillagos ég, még akkor is, ha tudjuk, hogy az aminosavak milyen sorrendben kapcsolódnak egymáshoz. A kutatók azóta küzdenek ezzel a problémával, hogy bebizonyították a sorrend és a struktúra közti kapcsolatot, ezen pedig az is csak mérsékelten segítette, amikor 1994-ben elindították a CASP-t.
A verseny során a szervezők minden második évben kiadnak száz olyan aminosav-szekvenciát, melyek struktúráját a kutatók már megállapították, de még nem hozták nyilvánosságra, a résztvevők feladata pedig az, hogy szoftverek segítségével megállapítsák a fehérjék alakját. A CASP-ra fejlesztett szoftverek jelentős részét már használják is a tudományban, egészen eddig azonban nem sikerült olyan megoldást találni, ami kiválthatná a költséges és időigényes laboratóriumi kísérletezést. A DeepMind 2018-ban jelent meg először a versenyen az AlphaFold eredeti változatával, és rögtön köröket is vert a versenytársakra, mostanra pedig felfoghatatlan előnyre tett szert. Az idei CASP-n a Google mesterséges intelligenciája
több tucatnyi fehérje struktúráját határozta meg 0,16 nanométeres, vagyis nagyjából egy atomnyi hibahatárral, ami nagyjából az a szint, amivel a laboratóriumi módszerek is operálnak.
Azokkal ellentétben ugyanakkor évekig tartó kísérletezés helyett néhány nap alatt megszületik az eredmény, ami hatalmas fegyvertény. Az új módszer pontos működése egyelőre nem ismert, először a héten ismertetik majd a CASP konferenciáján, ám az már most egyértelmű, hogy ennek segítségével a tudósok új gyógyszereket alkothatnak, még több betegség működését érthetik meg, hosszú távon pedig szintetikus fehérjék megalkotásához is felhasználhatják a technológiát. Az AlphaFold egyetlen hátránya a versenytársaival szemben az, hogy jóval lassabban dolgozik náluk – más algoritmusok ugyan jóval kevésbé pontosak, de pár másodperc alatt képesek eredményt produkálni, egyes esetekben pedig lehet, hogy pont ez a mérvadó. Az viszont így is egyértelmű, hogy ha a működési mechanizmusa nyilvánossá válik, az a tudományos kutatás új korszakát hozhatja el.
